Análise da estrutura horizontal de comunidades vegetais
A fitossociologia é uma área complexa das ciências naturais que tem como objetivo caracterizar a distribuição, o agrupamento e as relações entre espécies em comunidades vegetais naturais, de modo a descrever sua estrutura e composição. A análise fitossociológica permite caracterizar vegetações quanto a sua tipologia, estágio sucessional e estado de conservação, subsidiando o manejo, a recuperação e a conservação de ecossistemas.
Este é o primeiro de uma série de posts em que apresentarei procedimentos para estudos fitossociológicos empregando dados reais (acesse os outros posts em que apresento procedimentos para cálculo de índices ecológicos e construção de curvas de acumulação de espécies e de rarefação). Nesse exemplo, faremos a análise da estrutura horizontal de uma comunidade de cerrado. Começaremos importando e visualizando dados oriundos de um levantamento realizado a partir de 29 unidades amostrais de 100 m².
Obs: Para baixar os dados utilizados nesse exemplo, clique aqui.
# Importar base de dados
dados <- readxl::read_excel("dados_fitossociologia.xlsx")
# Visualizar dados importados
dados## # A tibble: 541 × 6
## Parcela Arvore DAP Altura Familia Especie
## <dbl> <dbl> <dbl> <dbl> <chr> <chr>
## 1 1 1 13.5 5 Dilleniaceae Curatella americana
## 2 1 2 5.57 3.5 Annonaceae Annona coriacea
## 3 1 3 5.09 4 Lauraceae Ocotea minarum
## 4 1 4 5.25 4 Lauraceae Ocotea minarum
## 5 1 5 11.8 4 Ebenaceae Diospyros hispida
## 6 1 6 7.96 5.5 Fabaceae Copaifera langsdorfii
## 7 1 7 8.91 4.5 Sapindaceae Matayba elaegnoides
## 8 1 8 5.89 3.5 Ebenaceae Diospyros hispida
## 9 1 9 8.75 4.5 Ebenaceae Diospyros hispida
## 10 1 10 4.77 2.5 Melastomataceae Miconia sp1
## # ℹ 531 more rows# Visualizar as espécies amostradas
unique(dados$Especie)## [1] "Curatella americana" "Annona coriacea"
## [3] "Ocotea minarum" "Diospyros hispida"
## [5] "Copaifera langsdorfii" "Matayba elaegnoides"
## [7] "Miconia sp1" "Pouteria torta"
## [9] "Byrsonima verbascifolia" "Alibertia edulis"
## [11] "Qualea grandiflora" "Leptolobium elegans"
## [13] "Myrcia sp1" "Terminalia argentea"
## [15] "Indeterminada 2" "Connarus suberosus"
## [17] "Xylopia aromatica" "Psidium laruotteanum"
## [19] "Andira cuiabensis" "Annona crassiflora"
## [21] "Dimorphandra mollis" "Indeterminada 1"
## [23] "Tabebuia aurea" "Campomanesia adamantium"
## [25] "Tachigali aurea" "Byrsonima basiloba"
## [27] "Qualea parviflora" "Schefflera macrocarpa"
## [29] "Tapirira guianensis" "Handroanthus albus"
## [31] "Rhamnidium elaeocarpum" "Anadenanthera falcata"
## [33] "Vochysia cinnamomea" "Byrsonima coccolobifolia"
## [35] "Couepia grandiflora" "Buchenavia tomentosa"
## [37] "Syagrus flexuosa" "Erythroxylum suberosum"
## [39] "Lafoensia pacari" "Eriotheca gracilipes"
## [41] "Magonia pubescens" "Kielmeyera coriacea"
## [43] "Bauhinia ungulata" "Vatairea macrocarpa"
## [45] "Byrsonima pachyphylla" "Roupala montana"
## [47] "Ouratea hexasperma" "Pouteria ramiflora"
## [49] "Dipteryx alata" "Eugenia aurata"
## [51] "Casearia sylvestris" "Machaerium acutifoium"
## [53] "Bauhinia rufa" "Eugenia punicifolia"
## [55] "Caryocar brasiliense" "Ouratea spectabilis"
## [57] "Himenaea courbaril" "Calliandra sp1"# Visualizar as famílias amostradas
unique(dados$Familia)## [1] "Dilleniaceae" "Annonaceae" "Lauraceae" "Ebenaceae"
## [5] "Fabaceae" "Sapindaceae" "Melastomataceae" "Sapotaceae"
## [9] "Malpighiaceae" "Rubiaceae" "Vochysiaceae" "Myrtaceae"
## [13] "Combretaceae" "Indeterminada" "Connaraceae" "Bignoniaceae"
## [17] "Araliaceae" "Anacardiaceae" "Rhamnaceae" "Chrysobalanaceae"
## [21] "Arecaceae" "Erythroxylaceae" "Lythraceae" "Malvaceae"
## [25] "Calophyllaceae" "Proteaceae" "Ochnaceae" "Salicaceae"
## [29] "Caryocaraceae"Ao todo 541 indivíduos foram amostrados no levantamento, abrangendo 58 espécies distribuídas em 29 famílias. Os principais parâmetros que descrevem a estrutura horizontal são apresentados a seguir.
| Parâmetro | Fórmula |
|---|---|
| Densidade absoluta | |
| Densidade relativa | |
| Frequência absoluta | |
| Frequência relativa | |
| Dominância absoluta | |
| Dominância relativa | |
| Índice de Valor de Importância |
Em que:
Agora vamos aplicar as fórmulas acima descritas aos dados que importamos para descrever a estrutura horizontal da comunidade amostrada. Ao final, geraremos uma tabela que apresenta os parâmetros calculados para todas as espécies, ordenadas pelo IVI.
# Definir tamanho da unidade amostral em m²
area_ua <- 100
# Número de parcelas
n_ua <- length(unique(dados$Parcela))
# Área total amostrada
A <- area_ua*n_ua
# Calcular área transversal dos indivíduos amostrados
dados$g <- pi*dados$DAP^2/40000
# Contar número de indivíduos por espécie
N <- table(dados$Especie)
# Densidades absoluta e relativa
DA <- N*10000/A
DR <- DA/sum(DA)*100
# Frequências absoluta e relativa
FA <- sapply(unique(dados$Especie),
function(x){
length(unique(subset(dados,dados$Especie==x)$Parcela))/n_ua
})*100
FA <- FA[order(names(FA))]
FR <- FA/sum(FA)*100
# Dominâncias absoluta e relativa
DOA <- aggregate(dados$g, list(dados$Especie), sum)$x*10000/A
DOR <- DOA/sum(DOA)*100
# Montar tabela
tabela_fito <- data.frame(Especie=names(N),
N=as.vector(N),
DA=round(as.vector(DA),1),
DR=round(as.vector(DR),1),
FA=round(as.vector(FA),1),
FR=round(as.vector(FR),1),
DoA=round(DOA,1),
DoR=round(DOR,1),
IVI=round(as.vector(DR+FR+DOR),1))
# Ordenar por IVI
tabela_fito <- tabela_fito[order(tabela_fito$IVI,decreasing = T),]
# Mostrar tabela
library(dplyr)
knitr::kable(tabela_fito, row.names = F)%>%
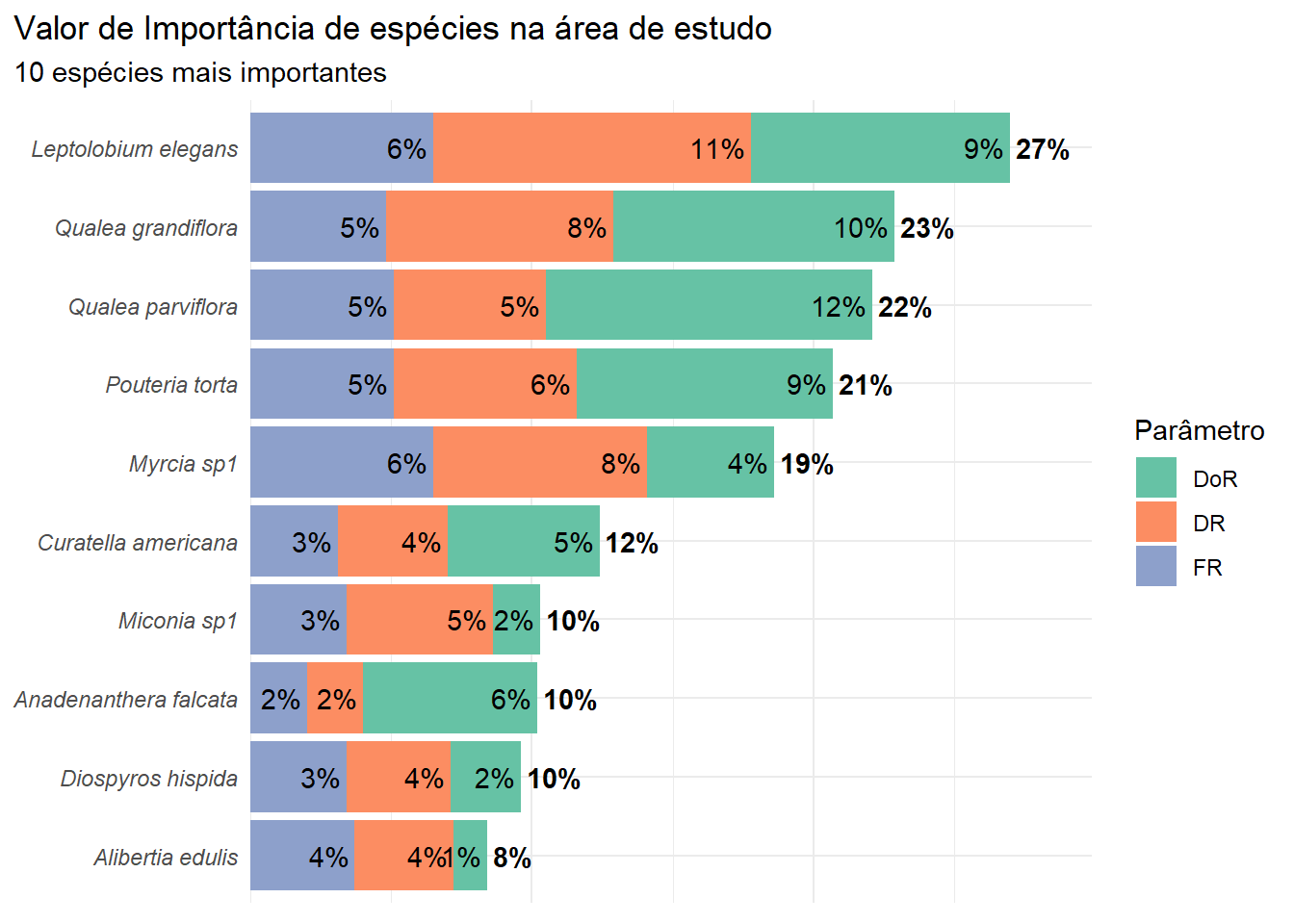
kableExtra::kable_styling(full_width = TRUE, position = "center",fixed_thead = T)| Especie | N | DA | DR | FA | FR | DoA | DoR | IVI |
|---|---|---|---|---|---|---|---|---|
| Leptolobium elegans | 61 | 210.3 | 11.3 | 65.5 | 6.5 | 1.7 | 9.2 | 27.0 |
| Qualea grandiflora | 44 | 151.7 | 8.1 | 48.3 | 4.8 | 1.8 | 10.0 | 22.9 |
| Qualea parviflora | 29 | 100.0 | 5.4 | 51.7 | 5.1 | 2.1 | 11.6 | 22.1 |
| Pouteria torta | 35 | 120.7 | 6.5 | 51.7 | 5.1 | 1.7 | 9.1 | 20.7 |
| Myrcia sp1 | 41 | 141.4 | 7.6 | 65.5 | 6.5 | 0.8 | 4.5 | 18.5 |
| Curatella americana | 21 | 72.4 | 3.9 | 31.0 | 3.1 | 1.0 | 5.4 | 12.3 |
| Anadenanthera falcata | 11 | 37.9 | 2.0 | 20.7 | 2.0 | 1.1 | 6.2 | 10.3 |
| Miconia sp1 | 28 | 96.6 | 5.2 | 34.5 | 3.4 | 0.3 | 1.7 | 10.3 |
| Diospyros hispida | 20 | 69.0 | 3.7 | 34.5 | 3.4 | 0.5 | 2.5 | 9.6 |
| Alibertia edulis | 19 | 65.5 | 3.5 | 37.9 | 3.7 | 0.2 | 1.2 | 8.5 |
| Matayba elaegnoides | 18 | 62.1 | 3.3 | 27.6 | 2.7 | 0.3 | 1.4 | 7.5 |
| Annona coriacea | 11 | 37.9 | 2.0 | 34.5 | 3.4 | 0.2 | 1.2 | 6.7 |
| Tabebuia aurea | 11 | 37.9 | 2.0 | 27.6 | 2.7 | 0.3 | 1.6 | 6.4 |
| Byrsonima coccolobifolia | 12 | 41.4 | 2.2 | 27.6 | 2.7 | 0.2 | 1.2 | 6.2 |
| Copaifera langsdorfii | 5 | 17.2 | 0.9 | 13.8 | 1.4 | 0.6 | 3.4 | 5.7 |
| Tapirira guianensis | 6 | 20.7 | 1.1 | 6.9 | 0.7 | 0.7 | 3.9 | 5.7 |
| Vochysia cinnamomea | 6 | 20.7 | 1.1 | 17.2 | 1.7 | 0.4 | 2.4 | 5.2 |
| Lafoensia pacari | 9 | 31.0 | 1.7 | 20.7 | 2.0 | 0.2 | 1.3 | 5.0 |
| Byrsonima verbascifolia | 8 | 27.6 | 1.5 | 24.1 | 2.4 | 0.2 | 1.0 | 4.9 |
| Eriotheca gracilipes | 7 | 24.1 | 1.3 | 20.7 | 2.0 | 0.3 | 1.5 | 4.8 |
| Byrsonima pachyphylla | 7 | 24.1 | 1.3 | 17.2 | 1.7 | 0.3 | 1.4 | 4.4 |
| Dimorphandra mollis | 6 | 20.7 | 1.1 | 17.2 | 1.7 | 0.3 | 1.5 | 4.3 |
| Tachigali aurea | 7 | 24.1 | 1.3 | 13.8 | 1.4 | 0.3 | 1.5 | 4.1 |
| Terminalia argentea | 5 | 17.2 | 0.9 | 13.8 | 1.4 | 0.3 | 1.8 | 4.1 |
| Connarus suberosus | 9 | 31.0 | 1.7 | 13.8 | 1.4 | 0.1 | 0.8 | 3.8 |
| Pouteria ramiflora | 7 | 24.1 | 1.3 | 20.7 | 2.0 | 0.1 | 0.5 | 3.8 |
| Handroanthus albus | 7 | 24.1 | 1.3 | 13.8 | 1.4 | 0.1 | 0.5 | 3.1 |
| Ocotea minarum | 7 | 24.1 | 1.3 | 13.8 | 1.4 | 0.1 | 0.4 | 3.0 |
| Machaerium acutifoium | 4 | 13.8 | 0.7 | 10.3 | 1.0 | 0.2 | 1.0 | 2.8 |
| Annona crassiflora | 4 | 13.8 | 0.7 | 13.8 | 1.4 | 0.1 | 0.6 | 2.7 |
| Bauhinia ungulata | 5 | 17.2 | 0.9 | 13.8 | 1.4 | 0.1 | 0.3 | 2.6 |
| Psidium laruotteanum | 4 | 13.8 | 0.7 | 13.8 | 1.4 | 0.1 | 0.5 | 2.6 |
| Campomanesia adamantium | 6 | 20.7 | 1.1 | 10.3 | 1.0 | 0.1 | 0.3 | 2.5 |
| Caryocar brasiliense | 4 | 13.8 | 0.7 | 10.3 | 1.0 | 0.1 | 0.7 | 2.5 |
| Buchenavia tomentosa | 4 | 13.8 | 0.7 | 10.3 | 1.0 | 0.1 | 0.5 | 2.3 |
| Xylopia aromatica | 4 | 13.8 | 0.7 | 13.8 | 1.4 | 0.0 | 0.2 | 2.3 |
| Magonia pubescens | 5 | 17.2 | 0.9 | 6.9 | 0.7 | 0.1 | 0.5 | 2.1 |
| Ouratea hexasperma | 4 | 13.8 | 0.7 | 10.3 | 1.0 | 0.1 | 0.3 | 2.1 |
| Roupala montana | 4 | 13.8 | 0.7 | 10.3 | 1.0 | 0.1 | 0.4 | 2.1 |
| Andira cuiabensis | 3 | 10.3 | 0.6 | 6.9 | 0.7 | 0.1 | 0.8 | 2.0 |
| Byrsonima basiloba | 2 | 6.9 | 0.4 | 6.9 | 0.7 | 0.2 | 0.8 | 1.9 |
| Kielmeyera coriacea | 3 | 10.3 | 0.6 | 10.3 | 1.0 | 0.1 | 0.3 | 1.9 |
| Indeterminada 1 | 3 | 10.3 | 0.6 | 3.4 | 0.3 | 0.1 | 0.8 | 1.7 |
| Schefflera macrocarpa | 2 | 6.9 | 0.4 | 6.9 | 0.7 | 0.1 | 0.6 | 1.6 |
| Vatairea macrocarpa | 2 | 6.9 | 0.4 | 6.9 | 0.7 | 0.1 | 0.4 | 1.4 |
| Erythroxylum suberosum | 2 | 6.9 | 0.4 | 6.9 | 0.7 | 0.0 | 0.2 | 1.3 |
| Syagrus flexuosa | 2 | 6.9 | 0.4 | 6.9 | 0.7 | 0.0 | 0.2 | 1.3 |
| Bauhinia rufa | 2 | 6.9 | 0.4 | 6.9 | 0.7 | 0.0 | 0.1 | 1.2 |
| Couepia grandiflora | 2 | 6.9 | 0.4 | 3.4 | 0.3 | 0.1 | 0.5 | 1.2 |
| Eugenia punicifolia | 2 | 6.9 | 0.4 | 6.9 | 0.7 | 0.0 | 0.1 | 1.2 |
| Ouratea spectabilis | 2 | 6.9 | 0.4 | 6.9 | 0.7 | 0.0 | 0.1 | 1.2 |
| Eugenia aurata | 2 | 6.9 | 0.4 | 3.4 | 0.3 | 0.0 | 0.3 | 1.0 |
| Calliandra sp1 | 2 | 6.9 | 0.4 | 3.4 | 0.3 | 0.0 | 0.1 | 0.8 |
| Indeterminada 2 | 1 | 3.4 | 0.2 | 3.4 | 0.3 | 0.1 | 0.3 | 0.8 |
| Dipteryx alata | 1 | 3.4 | 0.2 | 3.4 | 0.3 | 0.0 | 0.2 | 0.7 |
| Himenaea courbaril | 1 | 3.4 | 0.2 | 3.4 | 0.3 | 0.0 | 0.1 | 0.7 |
| Casearia sylvestris | 1 | 3.4 | 0.2 | 3.4 | 0.3 | 0.0 | 0.0 | 0.6 |
| Rhamnidium elaeocarpum | 1 | 3.4 | 0.2 | 3.4 | 0.3 | 0.0 | 0.0 | 0.6 |
Para finalizar, com uma abordagem mas sofisticada, vamos representar graficamente os resultados obtidos para as principais espécies.
library(dplyr)
library(tidyr)
library(forcats)
library(ggplot2)
# Quantidade de especies a serem mostradas no grafico
n_sp_plot <- 10
tabela_fito_long <- tabela_fito %>%
mutate(Especie = fct_reorder(Especie, IVI)) %>%
select(Especie, DR, FR, DoR) %>%
filter(Especie != 'NI') %>%
filter(row_number() <= n_sp_plot) %>%
pivot_longer(2:4, names_to = 'par', values_to = 'val')
# Plotar grafico
tabela_fito_long %>%
group_by(Especie) %>%
mutate(par = fct_reorder(par, c('DR', 'FR', 'DoR')),
IVI = if_else(par=='DoR', sum(val), NA_real_)) %>%
arrange(desc(par)) %>%
mutate(lab_y = cumsum(val)) %>%
ggplot(aes(x = Especie, fill = par))+
geom_bar(aes(y = val), stat = 'identity')+
geom_text(aes(y = lab_y, label = paste0(round(val), '%')), hjust = 1, nudge_y = -.2)+
geom_text(aes(y = IVI, label = paste0(round(IVI), '%')), fontface = 'bold', hjust = 0, nudge_y = .2)+
coord_flip()+
scale_y_continuous(expand=c(0,0), limits = function(x){c(0,max(x)*1.1)})+
scale_fill_brewer(name = 'Parâmetro', palette = 'Set2')+
labs(title = 'Valor de Importância de espécies na área de estudo',
subtitle = paste0(n_sp_plot, ' espécies mais importantes'))+
theme_minimal()+
theme(axis.title = element_blank(),
axis.line.x = element_blank(),
axis.text.x = element_blank(),
axis.ticks.x = element_blank(),
axis.text.y = element_text(face = 'italic'),
plot.title.position = 'plot')## Warning: Removed 20 rows containing missing values (`geom_text()`).