Cálculo de índices ecológicos
As análises fitossociológicas contemplam a utilização de índices ecológicos que exprimem a riqueza e a diversidade de comunidades vegetais, permitindo a comparação e o monitoramento dos recursos vegetais, além de fornecer subsídios ao seu manejo.
Nesse post calcularemos praticaremos o cálculo dos seguintes índices:
| Índice | Fórmula | Descrição |
|---|---|---|
| Índice de Margalef | Expressa riqueza de espécies | |
| Índice de Shannon | Expressa a heterogeneidade da composição florística ou o grau de incerteza em relação à espécie de um indivíduo amostrado ao acaso | |
| Índice de Simpson | Expressa a dominância de espécies e a probabilidade de dois indivíduos amostrados ao acaso pertencerem à mesma espécie | |
| Índice de Pielou | Exprime a equitabilidade, ou seja, o padrão de distribuição dos indivíduos entre as espécies. |
Em que:
Vamos às análises. Iniciaremos importando a mesma base de dados utilizada no post em que analisamos a estrutura horizontal e e também no post em que construímos curvas de acumulação de espécies e de rarefação. A seguir calcularemos todos os índices ecológicos acima apresentados, com auxílio do pacote vegan.
Obs: Para baixar os dados utilizados nesse exemplo, clique aqui.
# Importar dados
dados <- readxl::read_excel("dados_fitossociologia.xlsx")
# Visualizar dados
dados## # A tibble: 541 × 6
## Parcela Arvore DAP Altura Familia Especie
## <dbl> <dbl> <dbl> <dbl> <chr> <chr>
## 1 1 1 13.5 5 Dilleniaceae Curatella americana
## 2 1 2 5.57 3.5 Annonaceae Annona coriacea
## 3 1 3 5.09 4 Lauraceae Ocotea minarum
## 4 1 4 5.25 4 Lauraceae Ocotea minarum
## 5 1 5 11.8 4 Ebenaceae Diospyros hispida
## 6 1 6 7.96 5.5 Fabaceae Copaifera langsdorfii
## 7 1 7 8.91 4.5 Sapindaceae Matayba elaegnoides
## 8 1 8 5.89 3.5 Ebenaceae Diospyros hispida
## 9 1 9 8.75 4.5 Ebenaceae Diospyros hispida
## 10 1 10 4.77 2.5 Melastomataceae Miconia sp1
## # ℹ 531 more rows# Carregar pacotes necessários
library(vegan)
library(dplyr)
# Gerar matriz de frequência das espécies
matriz_freq <- xtabs(~Parcela+Especie, dados)
# Pacotes para geração da tabela em HTML
library(knitr)
library(kableExtra)
library(dplyr)
# Visualizar a matriz de frequência (cada linha é uma unidade amostral)
kable(matriz_freq[1:6,])%>%
kable_styling(fixed_thead = T)| Alibertia edulis | Anadenanthera falcata | Andira cuiabensis | Annona coriacea | Annona crassiflora | Bauhinia rufa | Bauhinia ungulata | Buchenavia tomentosa | Byrsonima basiloba | Byrsonima coccolobifolia | Byrsonima pachyphylla | Byrsonima verbascifolia | Calliandra sp1 | Campomanesia adamantium | Caryocar brasiliense | Casearia sylvestris | Connarus suberosus | Copaifera langsdorfii | Couepia grandiflora | Curatella americana | Dimorphandra mollis | Diospyros hispida | Dipteryx alata | Eriotheca gracilipes | Erythroxylum suberosum | Eugenia aurata | Eugenia punicifolia | Handroanthus albus | Himenaea courbaril | Indeterminada 1 | Indeterminada 2 | Kielmeyera coriacea | Lafoensia pacari | Leptolobium elegans | Machaerium acutifoium | Magonia pubescens | Matayba elaegnoides | Miconia sp1 | Myrcia sp1 | Ocotea minarum | Ouratea hexasperma | Ouratea spectabilis | Pouteria ramiflora | Pouteria torta | Psidium laruotteanum | Qualea grandiflora | Qualea parviflora | Rhamnidium elaeocarpum | Roupala montana | Schefflera macrocarpa | Syagrus flexuosa | Tabebuia aurea | Tachigali aurea | Tapirira guianensis | Terminalia argentea | Vatairea macrocarpa | Vochysia cinnamomea | Xylopia aromatica |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 4 | 0 | 3 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 2 | 0 | 0 | 0 | 4 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 6 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 4 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| 0 | 0 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 4 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| 0 | 0 | 1 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 7 | 0 | 1 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
# Número de indivíduos por parcela
n_ind_parc <- rowSums(matriz_freq)
# Número total de indivíduos amostrados
n_ind_tot <- sum(n_ind_parc)
# Número de espécies
n_sp_parc <- specnumber(matriz_freq)
# Número de espécies total
n_sp_tot <- specnumber(colSums(matriz_freq))
# Índice de Margalef para cada parcela
margalef_parc <- (n_sp_parc-1)/n_ind_parc
# Índice de Margalef para toda a comunidade
margalef_tot <- (n_sp_tot-1)/n_ind_tot
# Índice de Shannon para cada parcela
simpson_parc <- diversity(matriz_freq, index = 'simpson')
# Índice de Shannon para toda a comunidade
simpson_tot <- diversity(colSums(matriz_freq), index = 'simpson')
# Índice de Shannon para cada parcela
shannon_parc <- diversity(matriz_freq, index = 'shannon')
# Índice de Shannon para toda a comunidade
shannon_tot <- diversity(colSums(matriz_freq), index = 'shannon')
# Equabilidade de Pielou para cada parcela
pielou_parc <- shannon_parc/log(n_sp_parc)
# Equabilidade de Pielou para toda comunidade
pielou_tot <- shannon_tot/log(n_sp_tot)
# Gera tabela de resultados
tabela_indices <- data.frame(Parcela = names(margalef_parc),
Margalef = round(margalef_parc,2),
Simpson = round(simpson_parc,2),
Shannon = round(shannon_parc,2),
Pielou = round(pielou_parc,2))
tabela_indices <- bind_rows(tabela_indices,data.frame(Parcela = 'Total',
Margalef = round(margalef_tot,2),
Simpson = round(simpson_tot,2),
Shannon = round(shannon_tot,2),
Pielou = round(pielou_tot,2)))
knitr::kable(tabela_indices, row.names = FALSE)%>%
kableExtra::kable_styling(full_width = T, position = "center",fixed_thead = T)| Parcela | Margalef | Simpson | Shannon | Pielou |
|---|---|---|---|---|
| 1 | 0.44 | 0.84 | 1.93 | 0.93 |
| 2 | 0.47 | 0.84 | 1.93 | 0.93 |
| 3 | 0.47 | 0.83 | 2.01 | 0.87 |
| 4 | 0.47 | 0.86 | 2.11 | 0.92 |
| 5 | 0.53 | 0.84 | 2.03 | 0.92 |
| 6 | 0.44 | 0.79 | 1.87 | 0.85 |
| 7 | 0.33 | 0.68 | 1.36 | 0.84 |
| 8 | 0.57 | 0.85 | 2.04 | 0.93 |
| 9 | 0.78 | 0.86 | 2.04 | 0.98 |
| 10 | 0.79 | 0.91 | 2.44 | 0.98 |
| 11 | 0.48 | 0.88 | 2.38 | 0.90 |
| 12 | 0.46 | 0.83 | 2.19 | 0.85 |
| 13 | 0.53 | 0.88 | 2.23 | 0.93 |
| 14 | 0.40 | 0.86 | 2.06 | 0.94 |
| 15 | 0.53 | 0.88 | 2.20 | 0.96 |
| 16 | 0.63 | 0.91 | 2.48 | 0.97 |
| 17 | 0.42 | 0.76 | 1.78 | 0.81 |
| 18 | 0.58 | 0.92 | 2.66 | 0.96 |
| 19 | 0.47 | 0.74 | 1.83 | 0.80 |
| 20 | 0.50 | 0.81 | 2.27 | 0.82 |
| 21 | 0.45 | 0.87 | 2.21 | 0.92 |
| 22 | 0.50 | 0.75 | 1.49 | 0.93 |
| 23 | 0.56 | 0.81 | 1.74 | 0.97 |
| 24 | 0.43 | 0.87 | 2.16 | 0.94 |
| 25 | 0.50 | 0.80 | 1.70 | 0.95 |
| 26 | 0.56 | 0.84 | 2.10 | 0.91 |
| 27 | 0.52 | 0.90 | 2.40 | 0.94 |
| 28 | 0.30 | 0.79 | 1.98 | 0.80 |
| 29 | 0.50 | 0.89 | 2.35 | 0.95 |
| Total | 0.11 | 0.95 | 3.51 | 0.86 |
A tabela acima apesenta todos os parãmetros ecológicos calculados a nível de parcela e também para toda a comunidade.
Outro indicador ecológico comumente utilizado em estudos fitossociológicos é o Índice de Jaccard, que expressa a similaridade de espécies entre os locais amostrados, rendo a seguinte expressão matemática:
Em que:
# Índice de similaridade de Jaccard
indice_jaccard <- betadiver(matriz_freq,'j')Por fim, podemos analisar visualmente as relações de similaridade entre as parcelas ou adrupamento dessas por meio de um dendrograma. No gráfico abaixo podemos observar que as parcelas que, individualmente, apresentaram maior similaridade entre si foram 20 e 29.
# Gerar dendrograma
dendrograma <- hclust(indice_jaccard)
par(mfrow=c(1,1),mar=c(1,4,3,1))
plot(dendrograma, hang=1,
main = 'Dendrograma de similaridade',
ylab='Similaridade')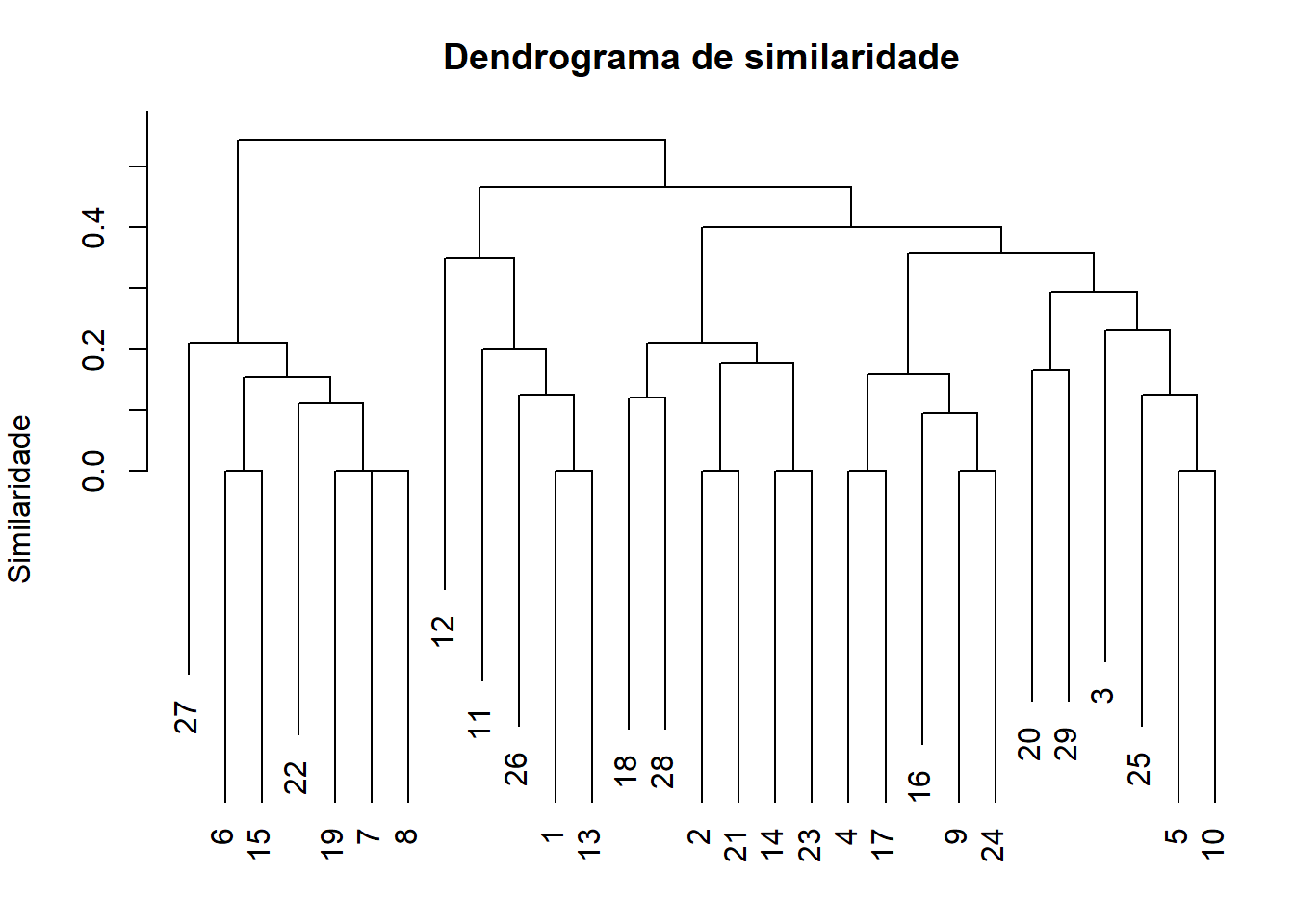 O dendrograma de similaridade é especialmente interessante para avaliar a a semelhança entre comunidades, embora possa também ser aplicado para comparar unidades amostrais em uma mesma comunidade. No presente exemplo, as unidades amostrais possuem área de 100 m², resultando em informações bastante limitadas a nível de parcela. Para este tipo de avaliação, unidades amostrais de maiores dimensões são mais adequadas pois reduzem a variação entre parcelas, permitindo uma avaliação mais abrangente.
O dendrograma de similaridade é especialmente interessante para avaliar a a semelhança entre comunidades, embora possa também ser aplicado para comparar unidades amostrais em uma mesma comunidade. No presente exemplo, as unidades amostrais possuem área de 100 m², resultando em informações bastante limitadas a nível de parcela. Para este tipo de avaliação, unidades amostrais de maiores dimensões são mais adequadas pois reduzem a variação entre parcelas, permitindo uma avaliação mais abrangente.