Funções de afilamento de forma fixa e variável
Na mensuração florestal, funções de afilamento de forma fixa são as mais difundidas devido à facilidade em seu ajuste e aplicação. O procedimento de integração exigido para obtenção dos volumes a diferentes alturas também é facilitado pelo uso desse tipo de função. Tradicionalmente, são mais utilizadas as funções polinomiais, como o modelo de Schöepfer (1966) e de Hradetzky (1976), descritos matematicamente pelas seguintes expressões.
Modelo de Schöepfer (1966)
Modelo de Hradetzky (1976)
em que:
As funções de afilamento de forma variável, introduzidas por Kozak (1988) e Newnham (1988), possuem como principal vantagem a capacidade de modelar diferentes formas de árvore em função de características dendrométricas, tornando possível a utilização de uma única equação de afilamento para árvores em diferentes idades e de diferentes portes. A utilização desta técnica permite a redução do número de funções de afilamento ajustados considerando povoamentos de uma mesma espécie estabelecidos em momentos e condições distintas. Neste exercício testaremos o modelo de Kozak (2004) e a função trigonométrica de Bi (2000) como representantes desta categoria de modelos.
Modelo de Kozak (2004)
Modelo de Bi (2000)
Para praticar o ajuste dos modelos, vamos importar um conjunto de dados de cubagens.
# Importar dados
library(readxl)
dados <- read_excel('dados_cubagens.xlsx')
dados## # A tibble: 54,415 × 7
## ARVORE DAP DI HI HT `HI/HT` `DI/DAP`
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 0.600 1.91 0.0735 1.47 0.05 3.18
## 2 1 0.600 1.59 0.1 1.47 0.0680 2.65
## 3 1 0.600 1.59 0.147 1.47 0.1 2.65
## 4 1 0.600 1.27 0.221 1.47 0.15 2.12
## 5 1 0.600 1.27 0.3 1.47 0.204 2.12
## 6 1 0.600 1.27 0.368 1.47 0.25 2.12
## 7 1 0.600 1.27 0.515 1.47 0.35 2.12
## 8 1 0.600 0.955 0.662 1.47 0.45 1.59
## 9 1 0.600 0.955 0.7 1.47 0.476 1.59
## 10 1 0.600 0.955 0.809 1.47 0.55 1.59
## # ℹ 54,405 more rowssummary(dados)## ARVORE DAP DI HI
## Min. : 1 Min. : 0.60 Min. : 0.000 Min. : 0.0735
## 1st Qu.: 840 1st Qu.:11.80 1st Qu.: 5.411 1st Qu.: 0.8315
## Median :1688 Median :18.10 Median :12.096 Median : 3.0600
## Mean :1688 Mean :18.39 Mean :13.455 Mean : 5.5745
## 3rd Qu.:2534 3rd Qu.:23.90 3rd Qu.:19.735 3rd Qu.: 8.8200
## Max. :3369 Max. :56.70 Max. :72.256 Max. :33.1300
## HT HI/HT DI/DAP
## Min. : 1.47 Min. :0.003018 Min. :0.0000
## 1st Qu.: 8.79 1st Qu.:0.074244 1st Qu.:0.4310
## Median :14.26 Median :0.350000 Median :0.8402
## Mean :14.23 Mean :0.396116 Mean :0.7572
## 3rd Qu.:19.40 3rd Qu.:0.750000 3rd Qu.:1.0459
## Max. :33.13 Max. :1.000000 Max. :3.8197# Plotar dispersão da relação hi/ht x di/dap
plot(`DI/DAP`~`HI/HT`, data = dados,
xlab = 'hi/ht', ylab = 'di/dap')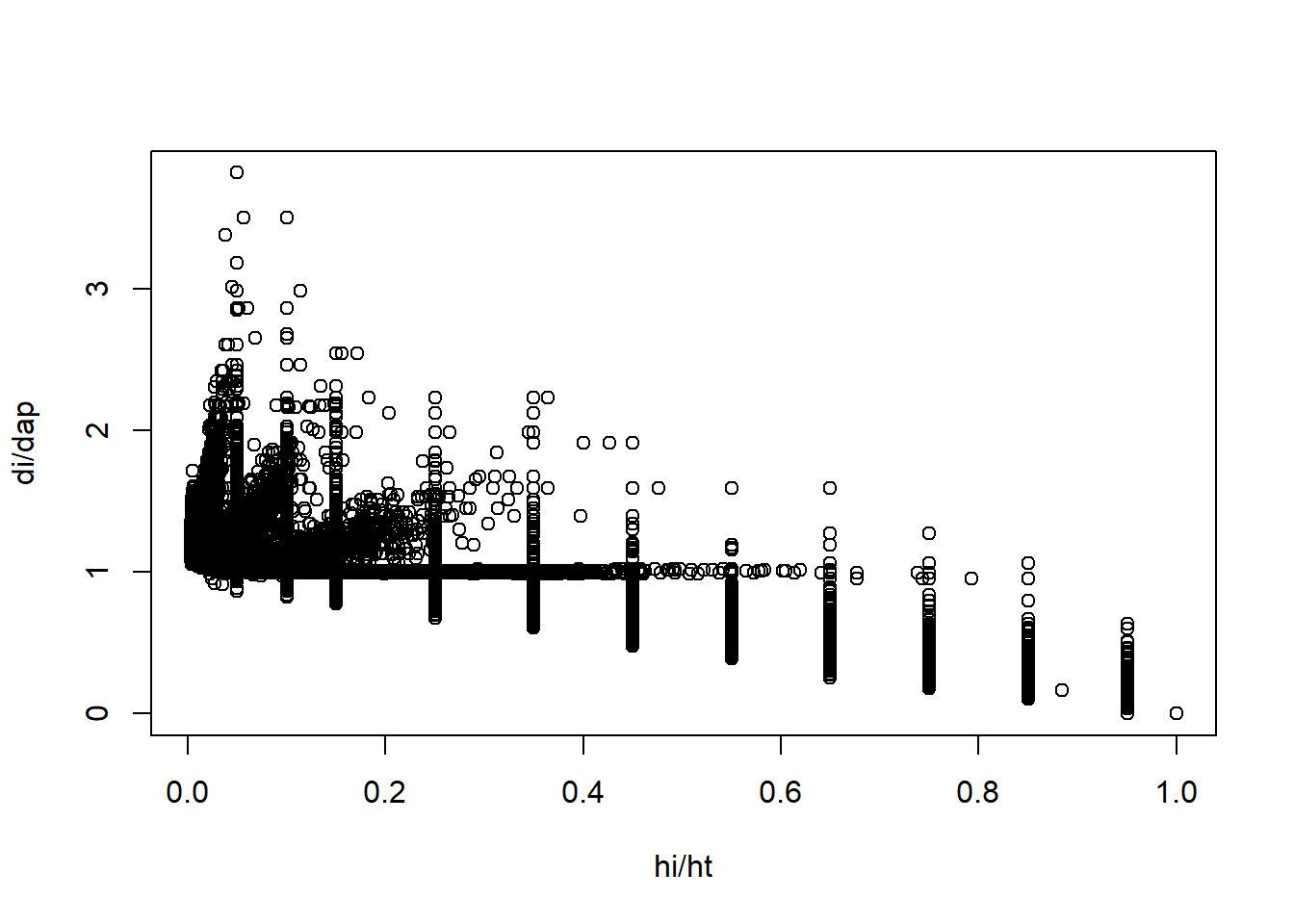
# Plotar dispersão da relação dap x ht
plot(HT~DAP, data = dados,
xlab = 'DAP (cm)', ylab = 'Ht (m)')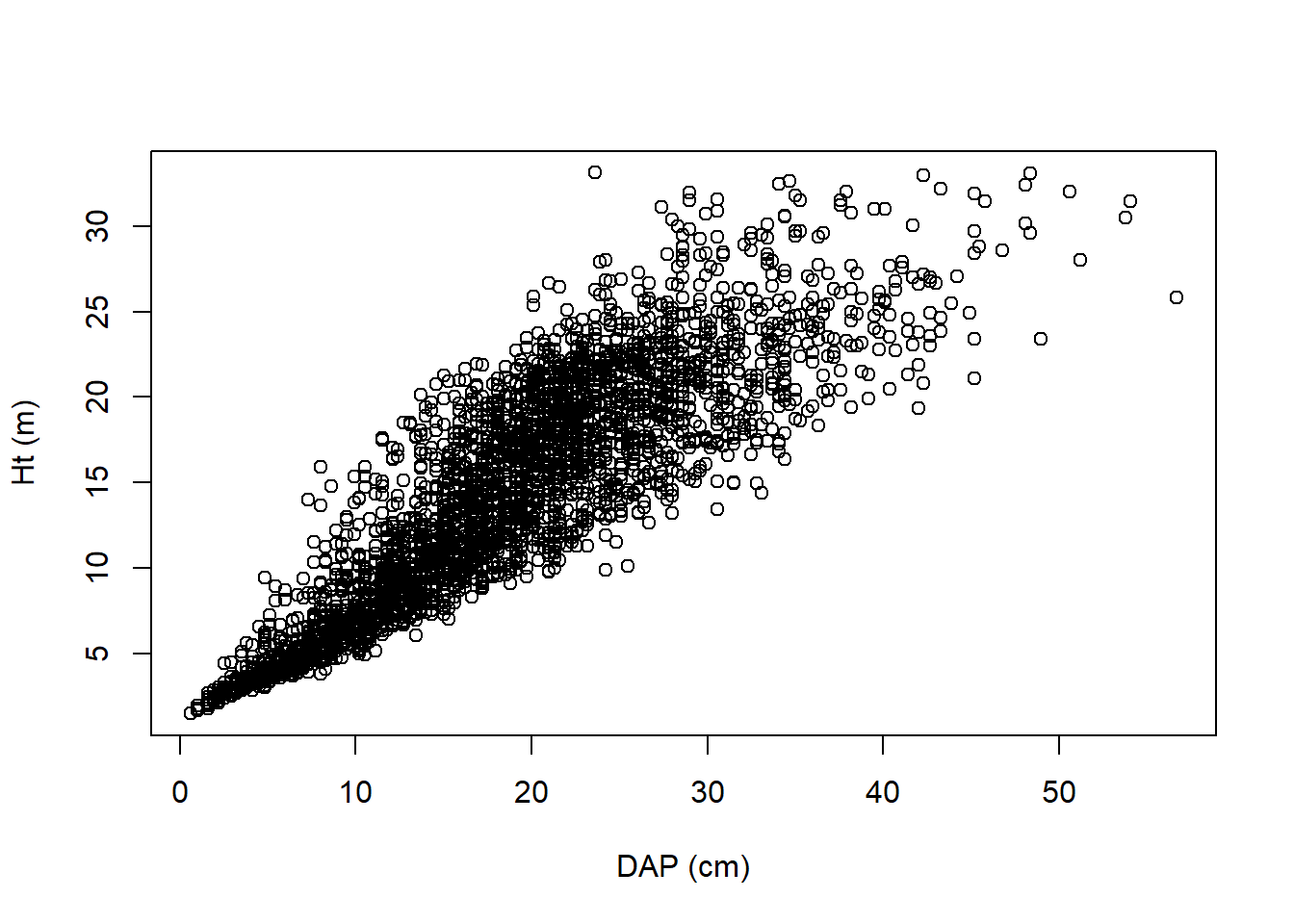
A base carregada consiste em dados de cubagens de 3.369 árvores, que tiveram diâmetros medidos a alturas relativas (5%, 10%, 15%, 25%, 35%, 45%, 55%, 65%, 75%, 85% e 95%), e também a alturas fixas (0,1, 0,3, 0,7 e 1,30 m).
Foram cubadas árvores de 0,6 a 56,7 cm de DAP, com alturas variando entre 1,47 e 33,13 m, indicando uma elevada variabilidade de portes de árvores.
Para utilização de funções polinomiais ou outras de forma fixa, é necessária a estratificação dos dados visando a redução da variabilidade existente. Neste caso, deverá ser gerada uma função de afilamento para cada estrato. Neste exemplo vamos estratificar o conjunto de dados a partir do DAP, sendo que o primeiro grupo compreenderá as árvores com menos de 5cm de dap e os demais serão formados com intervalos de 10 cm.
# Gerar as classes
dados$classe_dap <- cut(dados$DAP,
breaks = c(0,c(5,15,25,35,45,Inf)))
# Resumir as classes
summary(dados$classe_dap)## (0,5] (5,15] (15,25] (25,35] (35,45] (45,Inf]
## 2992 17072 22495 9744 1808 304Agora iremos ajustar o modelo de Schöepfer para cada um dos grupos.
# Criar uma lista para armazenar os modelos
funcoes_schoepfer <- list()
# Ajustar um modelo para cada grupo
for(i in levels(dados$classe_dap)){
funcoes_schoepfer[[i]] <- lm(`DI/DAP`~`HI/HT`+I(`HI/HT`^2)+I(`HI/HT`^3)+I(`HI/HT`^4)+I(`HI/HT`^5), data = subset(dados,dados$classe_dap==i))
}
# Resumo dos resultados dos modelos
lapply(funcoes_schoepfer, summary)## $`(0,5]`
##
## Call:
## lm(formula = `DI/DAP` ~ `HI/HT` + I(`HI/HT`^2) + I(`HI/HT`^3) +
## I(`HI/HT`^4) + I(`HI/HT`^5), data = subset(dados, dados$classe_dap ==
## i))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.66404 -0.13447 -0.03705 0.07904 2.09370
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.92582 0.02667 72.209 < 2e-16 ***
## `HI/HT` -4.70643 0.55981 -8.407 < 2e-16 ***
## I(`HI/HT`^2) 16.05552 3.46957 4.628 3.86e-06 ***
## I(`HI/HT`^3) -39.11067 8.78035 -4.454 8.72e-06 ***
## I(`HI/HT`^4) 43.79063 9.62348 4.550 5.57e-06 ***
## I(`HI/HT`^5) -17.91781 3.79077 -4.727 2.39e-06 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.2528 on 2986 degrees of freedom
## Multiple R-squared: 0.8186, Adjusted R-squared: 0.8183
## F-statistic: 2694 on 5 and 2986 DF, p-value: < 2.2e-16
##
##
## $`(5,15]`
##
## Call:
## lm(formula = `DI/DAP` ~ `HI/HT` + I(`HI/HT`^2) + I(`HI/HT`^3) +
## I(`HI/HT`^4) + I(`HI/HT`^5), data = subset(dados, dados$classe_dap ==
## i))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.36353 -0.06147 -0.01085 0.04006 0.87430
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.379487 0.003302 417.77 <2e-16 ***
## `HI/HT` -3.688856 0.077362 -47.68 <2e-16 ***
## I(`HI/HT`^2) 14.704332 0.517705 28.40 <2e-16 ***
## I(`HI/HT`^3) -36.228895 1.369753 -26.45 <2e-16 ***
## I(`HI/HT`^4) 39.094052 1.541516 25.36 <2e-16 ***
## I(`HI/HT`^5) -15.249265 0.617510 -24.70 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.1055 on 17066 degrees of freedom
## Multiple R-squared: 0.9396, Adjusted R-squared: 0.9396
## F-statistic: 5.313e+04 on 5 and 17066 DF, p-value: < 2.2e-16
##
##
## $`(15,25]`
##
## Call:
## lm(formula = `DI/DAP` ~ `HI/HT` + I(`HI/HT`^2) + I(`HI/HT`^3) +
## I(`HI/HT`^4) + I(`HI/HT`^5), data = subset(dados, dados$classe_dap ==
## i))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.23163 -0.03199 -0.00084 0.02766 0.48511
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.246046 0.001145 1087.90 <2e-16 ***
## `HI/HT` -4.071009 0.030005 -135.68 <2e-16 ***
## I(`HI/HT`^2) 17.476210 0.208070 83.99 <2e-16 ***
## I(`HI/HT`^3) -38.933380 0.561174 -69.38 <2e-16 ***
## I(`HI/HT`^4) 37.991111 0.639011 59.45 <2e-16 ***
## I(`HI/HT`^5) -13.708137 0.257950 -53.14 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.05219 on 22489 degrees of freedom
## Multiple R-squared: 0.981, Adjusted R-squared: 0.9809
## F-statistic: 2.316e+05 on 5 and 22489 DF, p-value: < 2.2e-16
##
##
## $`(25,35]`
##
## Call:
## lm(formula = `DI/DAP` ~ `HI/HT` + I(`HI/HT`^2) + I(`HI/HT`^3) +
## I(`HI/HT`^4) + I(`HI/HT`^5), data = subset(dados, dados$classe_dap ==
## i))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.20115 -0.03189 0.00065 0.02693 0.35102
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.216174 0.001565 776.87 <2e-16 ***
## `HI/HT` -4.202002 0.043583 -96.41 <2e-16 ***
## I(`HI/HT`^2) 18.609030 0.305483 60.92 <2e-16 ***
## I(`HI/HT`^3) -40.530337 0.825602 -49.09 <2e-16 ***
## I(`HI/HT`^4) 38.157955 0.940584 40.57 <2e-16 ***
## I(`HI/HT`^5) -13.252322 0.379751 -34.90 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.05069 on 9738 degrees of freedom
## Multiple R-squared: 0.9816, Adjusted R-squared: 0.9815
## F-statistic: 1.036e+05 on 5 and 9738 DF, p-value: < 2.2e-16
##
##
## $`(35,45]`
##
## Call:
## lm(formula = `DI/DAP` ~ `HI/HT` + I(`HI/HT`^2) + I(`HI/HT`^3) +
## I(`HI/HT`^4) + I(`HI/HT`^5), data = subset(dados, dados$classe_dap ==
## i))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.142770 -0.027460 -0.002403 0.022018 0.257885
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.200521 0.003163 379.56 <2e-16 ***
## `HI/HT` -4.266131 0.091633 -46.56 <2e-16 ***
## I(`HI/HT`^2) 18.779570 0.647795 28.99 <2e-16 ***
## I(`HI/HT`^3) -39.872610 1.753872 -22.73 <2e-16 ***
## I(`HI/HT`^4) 36.363012 1.998715 18.19 <2e-16 ***
## I(`HI/HT`^5) -12.208106 0.806897 -15.13 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.0463 on 1802 degrees of freedom
## Multiple R-squared: 0.9846, Adjusted R-squared: 0.9845
## F-statistic: 2.303e+04 on 5 and 1802 DF, p-value: < 2.2e-16
##
##
## $`(45,Inf]`
##
## Call:
## lm(formula = `DI/DAP` ~ `HI/HT` + I(`HI/HT`^2) + I(`HI/HT`^3) +
## I(`HI/HT`^4) + I(`HI/HT`^5), data = subset(dados, dados$classe_dap ==
## i))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.122237 -0.031020 0.003504 0.025404 0.166978
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.191807 0.007745 153.874 < 2e-16 ***
## `HI/HT` -4.527408 0.231443 -19.562 < 2e-16 ***
## I(`HI/HT`^2) 20.094868 1.650086 12.178 < 2e-16 ***
## I(`HI/HT`^3) -42.661480 4.478459 -9.526 < 2e-16 ***
## I(`HI/HT`^4) 38.969213 5.107729 7.629 3.20e-13 ***
## I(`HI/HT`^5) -13.070928 2.062564 -6.337 8.63e-10 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.04846 on 298 degrees of freedom
## Multiple R-squared: 0.9834, Adjusted R-squared: 0.9832
## F-statistic: 3539 on 5 and 298 DF, p-value: < 2.2e-16Para ajustar o modelo de Kozak, vamos utilizar a função nlsLM do pacote minpack.LM. Esta função busca parâmetros de modelos não lineares empregando o algoritmo Levenberg-Marquadt e costuma ser mais eficiente que a função nls, especialmente quando buscamos ajustar modelos mais complexos.
library(minpack.lm)
# Ajuste do modelo de Kozak
kozak <- nlsLM(DI~b0*(DAP^b1)*((1-(HI/HT)^(1/4))/(1-p^(1/4)))^(b2+b3*(1/exp(DAP/HT))+b4*DAP^((1-(HI/HT)^(1/4))/(1-p^(1/4)))+b5*((1-(HI/HT)^(1/4))/(1-p^(1/4)))^(DAP/HT)),
data = dados,
start=list(b0=1,b1=1,b2=1,b3=-1,b4=0.00001,b5=-0.01, p = 0.3))
summary(kozak)##
## Formula: DI ~ b0 * (DAP^b1) * ((1 - (HI/HT)^(1/4))/(1 - p^(1/4)))^(b2 +
## b3 * (1/exp(DAP/HT)) + b4 * DAP^((1 - (HI/HT)^(1/4))/(1 -
## p^(1/4))) + b5 * ((1 - (HI/HT)^(1/4))/(1 - p^(1/4)))^(DAP/HT))
##
## Parameters:
## Estimate Std. Error t value Pr(>|t|)
## b0 1.129e+00 6.062e-03 186.26 <2e-16 ***
## b1 8.896e-01 9.135e-04 973.88 <2e-16 ***
## b2 6.994e-01 2.313e-03 302.40 <2e-16 ***
## b3 -6.180e-01 6.870e-03 -89.95 <2e-16 ***
## b4 2.752e-06 3.284e-07 8.38 <2e-16 ***
## b5 -3.390e-02 9.240e-04 -36.69 <2e-16 ***
## p 2.868e-01 4.167e-03 68.83 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.248 on 54408 degrees of freedom
##
## Number of iterations to convergence: 10
## Achieved convergence tolerance: 1.49e-08#Ajuste do modelo de Bi
bi = nlsLM(DI ~ DAP*(log(sin((pi/2)*(HI/HT)))/(log(sin((pi/2)*(1.3/HT)))))**
(b0+b1*sin((pi/2)*(HI/HT))+b2*cos((3*pi/2)*(HI/HT))+b3*(sin((pi/2)*(HI/HT))/(HI/HT))+
b4*DAP+b5*(HI/HT)*DAP**0.5+b6*(HI/HT)*HT**0.5),
data=dados,
start=c(b0=1.8,b1=-0.2,b2=-0.04,b3=-0.9,b4=-0.0006,b5=0.07,b6=-.14))
summary(bi)##
## Formula: DI ~ DAP * (log(sin((pi/2) * (HI/HT)))/(log(sin((pi/2) * (1.3/HT)))))^(b0 +
## b1 * sin((pi/2) * (HI/HT)) + b2 * cos((3 * pi/2) * (HI/HT)) +
## b3 * (sin((pi/2) * (HI/HT))/(HI/HT)) + b4 * DAP + b5 * (HI/HT) *
## DAP^0.5 + b6 * (HI/HT) * HT^0.5)
##
## Parameters:
## Estimate Std. Error t value Pr(>|t|)
## b0 1.801e+00 1.212e-02 148.65 <2e-16 ***
## b1 -2.803e-01 6.123e-03 -45.78 <2e-16 ***
## b2 -5.107e-02 1.565e-03 -32.62 <2e-16 ***
## b3 -9.142e-01 7.134e-03 -128.14 <2e-16 ***
## b4 -8.873e-04 5.188e-05 -17.10 <2e-16 ***
## b5 7.406e-02 1.161e-03 63.79 <2e-16 ***
## b6 -1.277e-01 9.892e-04 -129.13 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.9302 on 54408 degrees of freedom
##
## Number of iterations to convergence: 4
## Achieved convergence tolerance: 1.49e-08Agora que temos as funções de forma fixa e variável ajustadas, vamos comparar os métodos. Para tal, precisamos armazenar os resíduos em vetores e calcular o erro quadrático médio em relação ao
# Criar conjuntos que armazenarão os resultados das funções de Schoepfer
di_schoepfer <- c()
residuos_schoepfer <- c()
# Calcular os resíduos a partir das funções de Schoepfer
for(i in unique(dados$classe_dap)){
di_schoepfer <- append(di_schoepfer,predict(funcoes_schoepfer[[i]])*subset(dados$DAP,dados$classe_dap==i))
residuos_schoepfer <- append(residuos_schoepfer,subset(dados$DI,dados$classe_dap==i)-predict(funcoes_schoepfer[[i]])*subset(dados$DAP,dados$classe_dap==i))}
# Resíduos da função de Kozak e Bi
residuos_kozak <- residuals(kozak)
residuos_bi <- residuals(bi)
# Calcular o erro quadrático médio
eqm_schoepfer <- sqrt(sum(residuos_schoepfer^2)/nrow(dados))
eqm_kozak <- sqrt(sum(residuos_kozak^2)/nrow(dados))
eqm_bi <- sqrt(sum(residuos_bi^2)/nrow(dados))
# Calcular o erro quadrático médio percentual
eqm_perc_schoepfer <- eqm_schoepfer/mean(dados$DI)*100
eqm_perc_kozak <- eqm_kozak/mean(dados$DI)*100
eqm_perc_bi <- eqm_bi/mean(dados$DI)*100
# Plotar resultados
par(mfrow=c(1,3))
plot(residuos_schoepfer~di_schoepfer,
main = paste('Schoepfer - EQM% = ', round(eqm_perc_schoepfer,2)),
xlab = 'Di estimado', ylab = 'Resíduos',
ylim = c(-15,15))+
lines(x=c(0,70),y=c(0,0))
plot(residuos_kozak~dados$DI,
main = paste('Kozak - EQM% = ', round(eqm_perc_kozak,2)),
xlab = 'Di estimado', ylab = 'Resíduos',
ylim = c(-15,15))+
lines(x=c(0,70),y=c(0,0))
plot(residuos_bi~dados$DI,
main = paste('Bi - EQM% = ', round(eqm_perc_bi,2)),
xlab = 'Di estimado', ylab = 'Resíduos',
ylim = c(-15,15))+
lines(x=c(0,70),y=c(0,0))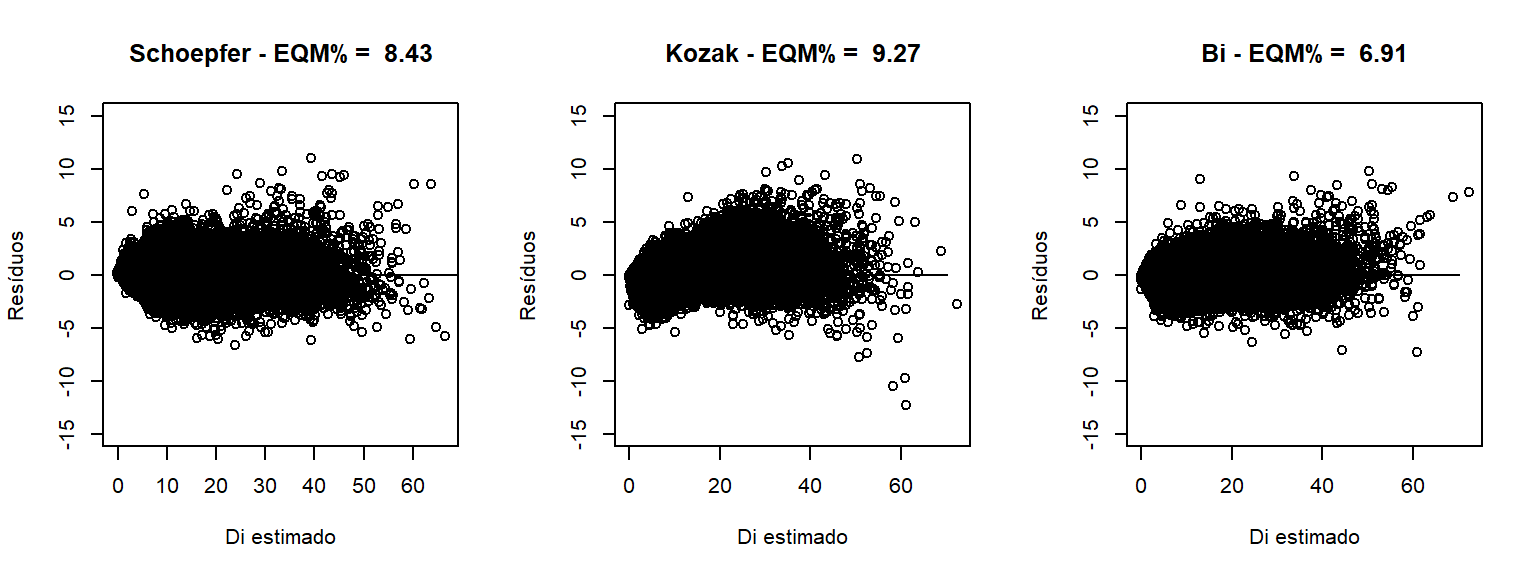 O modelo trigonométrico de Bi superou os demais tanto pelo menor erro quadrático médio, quanto pela dispersão de resíduos mais homogeneamente distribuída ao longo da linha de ajuste.
O modelo trigonométrico de Bi superou os demais tanto pelo menor erro quadrático médio, quanto pela dispersão de resíduos mais homogeneamente distribuída ao longo da linha de ajuste.
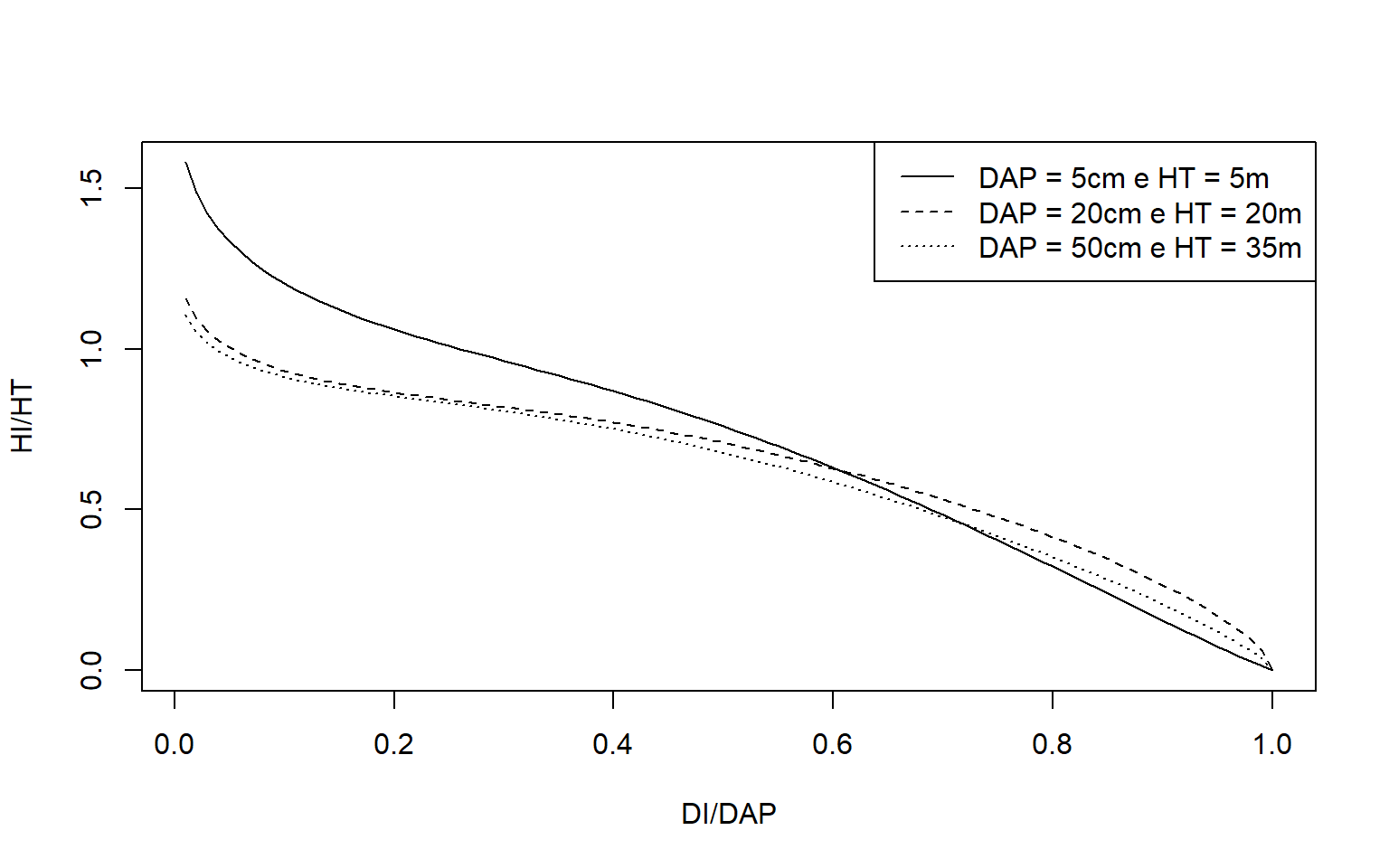
Por fim, vamos visualizar a capacidade da equação gerada a partir do modelo de Bi em descrever diferentes formas de fuste.
# Simular dados de árvores de diferentes portes
arvores_simuladas <- data.frame(DAP = rep(c(5,20,50),each = 100),
HT = rep(c(5,25,35),each = 100),
`HI/HT` = seq(0.01,1,0.01))
arvores_simuladas$HI <- arvores_simuladas$HI.HT*arvores_simuladas$HT
# Aplicar a equação ajustada de Bi aos dados simulados
arvores_simuladas$DI <- predict(bi,arvores_simuladas)
# Plotar curvas de afilamento
lt <- 1
plot(I(DI/DAP)~I(HI/HT),subset(arvores_simuladas, arvores_simuladas$DAP==unique(arvores_simuladas$DAP)[1]), type = 'l', lty = lt,
xlab ='DI/DAP', ylab = 'HI/HT')+
for (i in unique(arvores_simuladas$DAP)[-1]) {
lt <- lt+1
lines(I(DI/DAP)~I(HI/HT),
subset(arvores_simuladas,arvores_simuladas$DAP==i), lty = lt)
}
legend('topright', legend= c('DAP = 5cm e HT = 5m', 'DAP = 20cm e HT = 20m', 'DAP = 50cm e HT = 35m'),
lty = 1:3)