Ilustrando o perfil esquemático de uma comunidade florestal
Para caracterização da tipologia, do estado de conservação e do estágio sucessional de comunidades florestais, são utilizados diversos parâmetros fitossociológicos como densidade, frequência, dominância, valor de importância e índices de diversidade. Tais indicadores permitem descrever detalhadamente a fitofisionomia dos locais de estudo, conforme demonstrado em postagens anteriores sobre este tema.
Ainda que a utilização de todos estes parâmetros permita uma adequada descrição, estes não passam de abstrações teóricas que buscam representar as comunidades vegetais por meio de números, tabelas e gráficos.
Buscando uma representação mais fiel à realidade do observador (i.e. aquele que realiza o levantamento em campo), alguns ecólogos e estudiosos da fitossociologia buscam representar o perfil esquemático das florestas por meio de ilustrações feitas à mão - habilidade restrita àqueles que possuem este talento.
Nesta postagem ofereço um código que permite representar o perfil esquemático de uma comunidade florestal utilizando recursos do pacote ggplot. O mais interessante é que o procedimento aloca as árvores no perfil de maneira probabilística, em função dos dados coletados em campo.
Vou explicar. Começamos importando os dados.
Obs: Para baixar os dados utilizados nesse exemplo, clique aqui.
# Importar base de dados
dados <- readxl::read_excel("dados_levantamento_fitossociologico.xlsx")
head(dados)## # A tibble: 6 × 8
## Parcela Gen_esp Gen_esp_abv Família CAP DAP H `Estrato vertical`
## <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <chr>
## 1 1 Prunus brasi… P. brasili… Rosace… 46.5 14.8 12 Intermediário
## 2 1 Nectandra la… N. lanceol… Laurac… 106 33.7 17 Superior
## 3 1 Dalbergia fr… D. frutesc… Fabace… 24 7.64 17 Superior
## 4 1 Myrcia splen… M. splende… Myrtac… 18.5 5.89 7 Sub-bosque
## 5 1 Myrcia splen… M. splende… Myrtac… 21 6.68 9 Intermediário
## 6 1 Maytenus evo… M. evonymo… Celast… 46 14.6 10 IntermediárioOs dados são oriundos de 30 unidades amostrais medidas em uma fragmento de Floresta Ombrófila Mista (mata de araucária) no Paraná. Para cada indivíduo amostrado nas parcelas, foram medidas a CAP (circunferência à altura do peito) e a altura total, além da determinação do epíteto botânico e do estrato vertical em que se encontrava no momento do levantamento.
O procedimento basicamente se dá nos seguintes passos:
1. Obter altura e DAP médios para cada espécie em cada estrato;
2. Contabilizar número de ocorrências de cada espécie em cada estrato;
3. Contabilizar proporção de indivíduos em cada estrato;
4. Gerar indivíduos a serem ilustrados sorteando a espécie e o estrato, com base em probabilidades geradas a partir dos dados coletados;
5. Plotar os indivíduos sorteados considerando os valores de altura e diâmetro médios de cada espécie em cada estrato.
Traduzindo para o R, o procedimento é o seguinte.
perfil_esquematico <- function(narv){
# Carregar pacote de manipulação de dados
library(dplyr)
# Obter altura média para cada espécie em cada estrato vertical
dados_perfil <- dados %>% group_by(`Estrato vertical`,Gen_esp_abv) %>% summarise(mean(H)) %>%
# Renamoear colunas
rename('estrato'=1, 'especie'=2,'h.med'=3) %>%
ungroup() %>%
# Obter DAP médio para cada espécie em cada estrato vertical
mutate(dap= group_by(dados, `Estrato vertical`, Gen_esp_abv) %>% summarise(dap=mean(DAP)) %>% pull(dap)) %>%
# Obter abundância para cada espécie em cada estrato vertical
mutate(ab= group_by(dados, `Estrato vertical`, Gen_esp_abv) %>% summarise(ab=n()) %>% pull(ab))
# Separar dados por estrato
emergente <- dados_perfil %>% filter(estrato == 'Emergente')
subbosque <- dados_perfil %>% filter(estrato == 'Sub-bosque')
superior <- dados_perfil %>% filter(estrato == 'Superior')
intermediario <- dados_perfil %>% filter(estrato == 'Intermediário')
# Contar árvores por estrato
narv_estrato <- round(table(dados$`Estrato vertical`)/sum(table(dados$`Estrato vertical`))*narv,0)
# Corrigir diferença, se existente, entre o argumento `narv` e o número de árvres sorteadas
if(sum(narv_estrato)!=narv){
narv_estrato[which(narv_estrato==max(narv_estrato))] <- narv_estrato[which(narv_estrato==max(narv_estrato))]+1
}
# Probabilidade de ocorrência das espécies em cada estrato
emergente$prob <- emergente$ab/sum(emergente$ab)
subbosque$prob <- subbosque$ab/sum(subbosque$ab)
superior$prob <- superior$ab/sum(superior$ab)
intermediario$prob <- intermediario$ab/sum(intermediario$ab)
# Sortear árvores por estrato
sample_eme <- tryCatch({sample(1:nrow(emergente), narv_estrato[1], replace = TRUE, emergente$prob)},
error = function (e) {0}
)
sample_int <- tryCatch({
sample(1:nrow(intermediario), narv_estrato[2], replace = TRUE, intermediario$prob)},
error = function (e) {0}
)
sample_sub <- tryCatch({
sample_sub <- sample(1:nrow(subbosque), narv_estrato[3], replace = TRUE, subbosque$prob)},
error = function (e) {0}
)
sample_sup <- tryCatch({
sample(1:nrow(superior), narv_estrato[4], replace = TRUE, superior$prob)},
error = function (e) {0}
)
sample_eme <- emergente[sample_eme,]
sample_int <- intermediario[sample_int,]
sample_sub <- subbosque[sample_sub,]
sample_sup <- superior[sample_sup,]
# Unir estratos da floresta simulada
sample <- bind_rows(sample_eme,sample_int,sample_sub,sample_sup) %>%
mutate(dap01=(dap-min(dap))/(max(dap)-min(dap))*(.6-.1)+.1,
id = sample(1:narv,narv))
id_sp <- unique(sample$especie)
sample$id_sp <- match(sample$especie,id_sp)
# Carregar pacotes necessários
library(ggplot2)
library(ggforce)
library(ggrepel)
# Plotar floresta
perfil <- ggplot(sample)+
geom_bar(aes(x=id,y=h.med, width=dap01),stat = 'identity', color = 'black', fill = 'burlywood4')+
geom_ellipse(aes(x0=id, y0=h.med,
a=1+3*dap01*h.med/(1/narv*500), #função genérica de diâmetro de copa
b=1,angle=0), fill = 'darkgreen')+
geom_label_repel(aes(x=id,y=h.med+.5,label=especie), direction = 'y', fontface = 'bold', size = 2, alpha=.8, segment.alpha = 0)+
scale_x_continuous(breaks = 1:narv, expand = c(.01,0))+
scale_y_continuous(expand=c(0,0), limits = c(0,max(sample$h.med)+5))+
xlab('')+
ylab('Altura (m)')+
theme_bw()+
theme(panel.grid = element_blank(),
axis.text.x = element_blank(),
axis.ticks.x = element_blank())
return(perfil)
}A função acima possui como único parâmetro o número de árvores a serem apresentadas. Vamos definir uma quantidade, e executar a função.
# Número de árvores a serem representadas plotar
quantidade_de_arvores <- 30
# Executar função
perfil_esquematico(quantidade_de_arvores)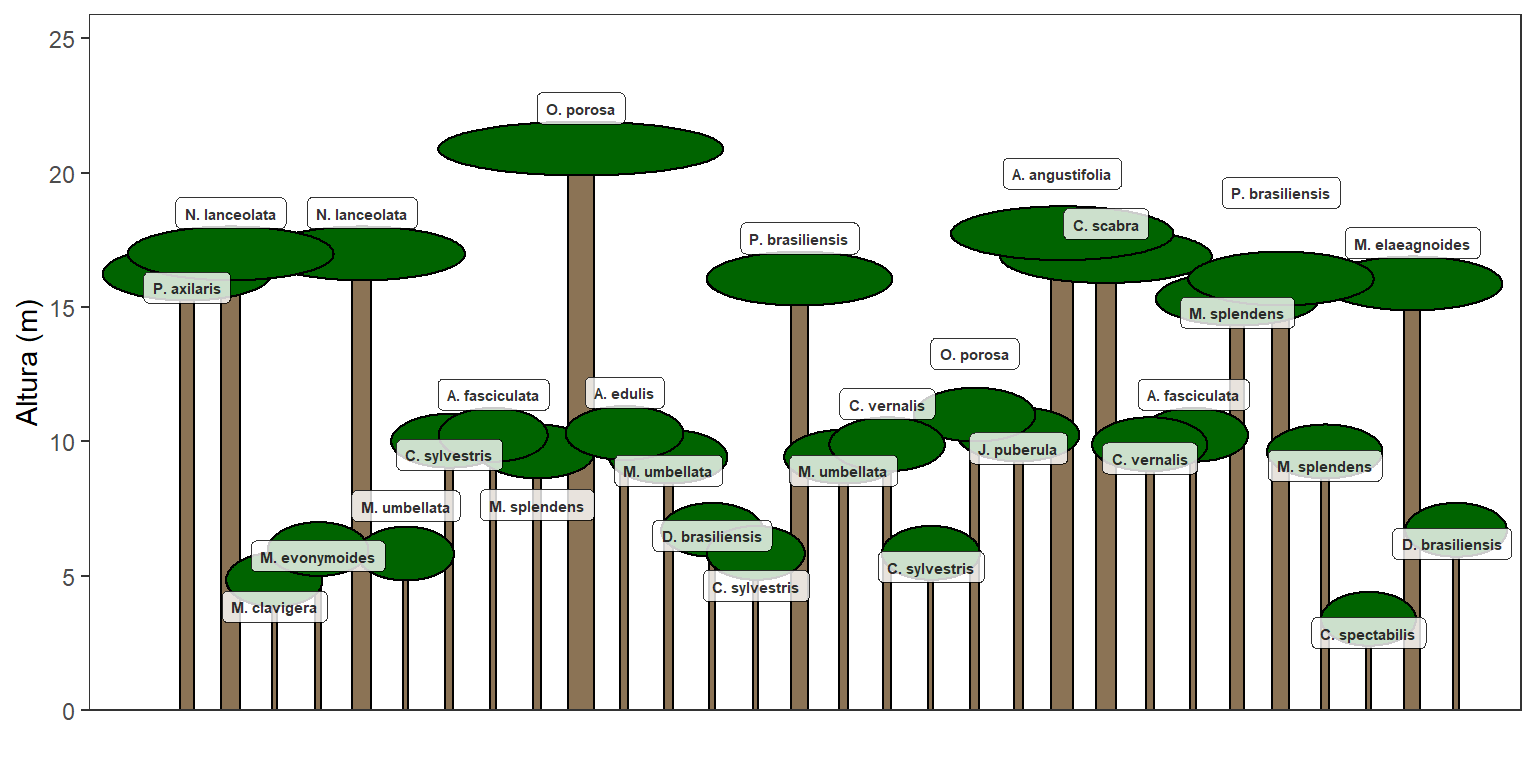 Como a ilustração é criada em função de probabilidades de ocorrência das espécies em cada estrato, se executarmos a função novamente utilizando o mesmo parâmetro, teremos uma nova representação.
Como a ilustração é criada em função de probabilidades de ocorrência das espécies em cada estrato, se executarmos a função novamente utilizando o mesmo parâmetro, teremos uma nova representação.
# Executar função
perfil_esquematico(quantidade_de_arvores)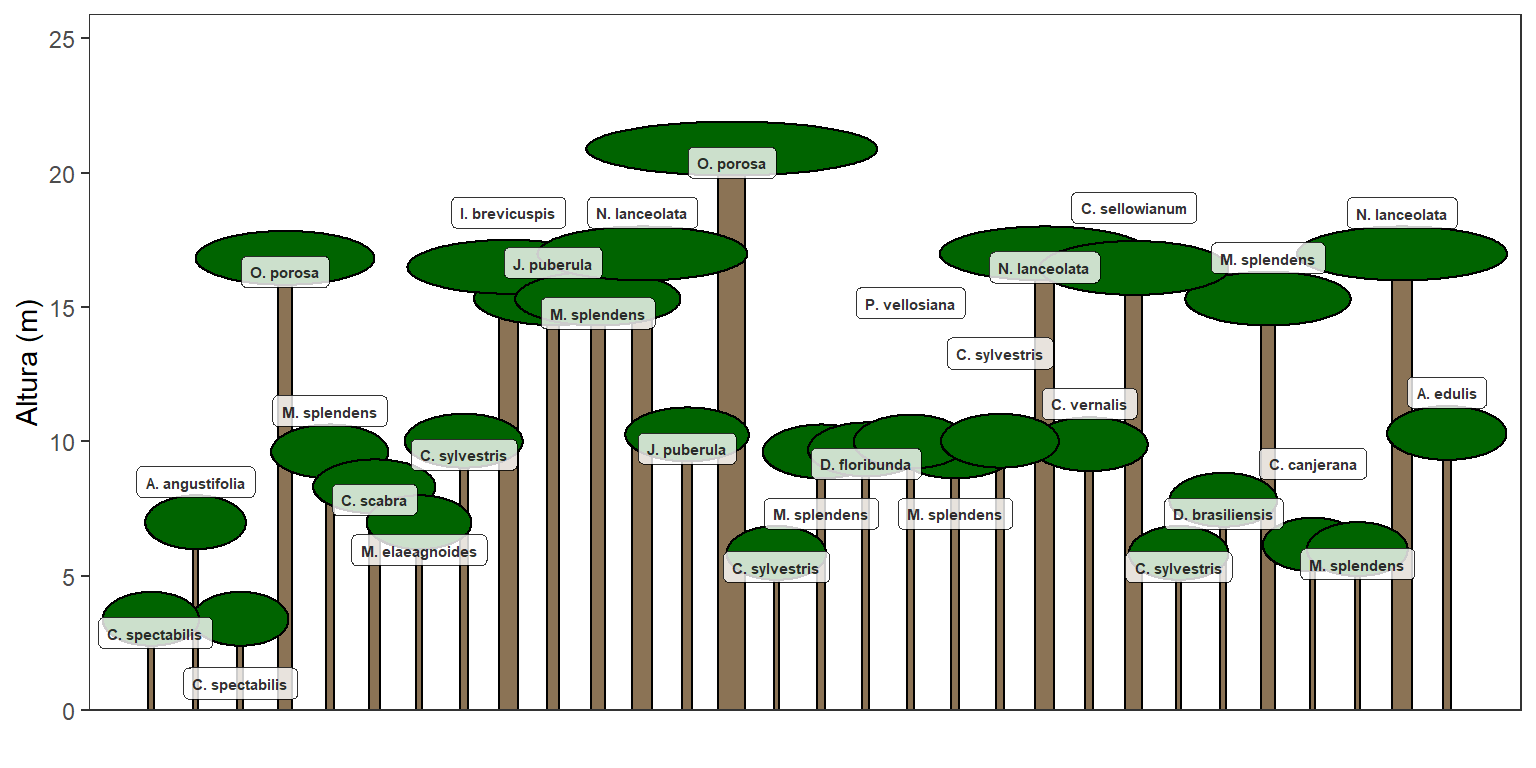 Podemos também variar a quantidade de indivíduos.
Podemos também variar a quantidade de indivíduos.
# Executar função
perfil_esquematico(20)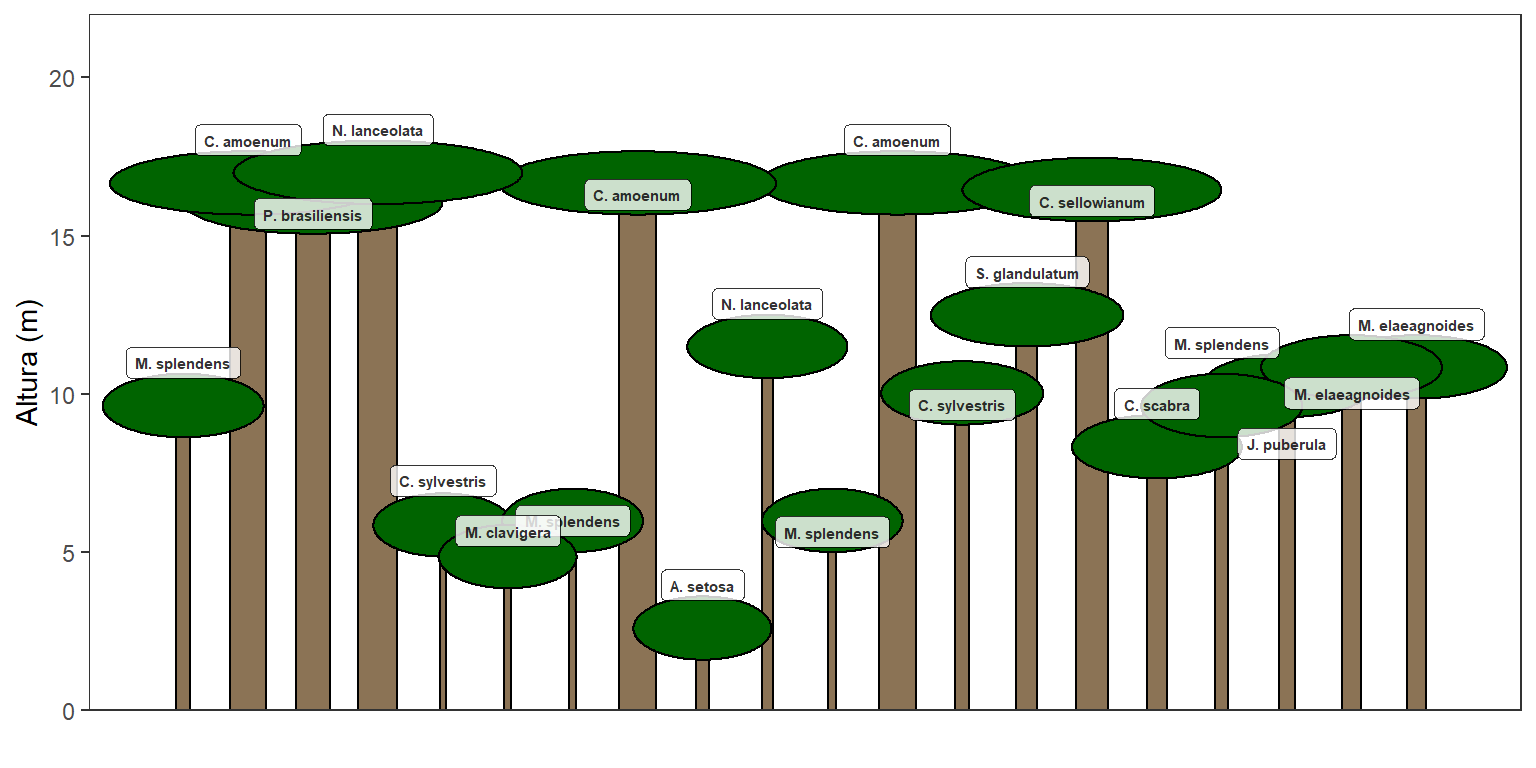 Como o procedimento de sorteio arredonda o número de árvores a serem sorteadas em cada estrato vertical (afinal não temos como sortear 1,5 árvores) a função pode retornar um erro caso quantidade total de árvores sorteadas seja inferior ao número declarado na função.
A função pode ser customizada de inúmeras maneiras. Utilizei um cálculo genérico para gerar o diâmetro das copas pois não possuo esses registros na base de dados utilizada nesse exemplo, no entanto, altura e diâmetro de copa, se coletados, podem ser utilizados para dimensionar as elipses que representam as copas das árvores. A função
Como o procedimento de sorteio arredonda o número de árvores a serem sorteadas em cada estrato vertical (afinal não temos como sortear 1,5 árvores) a função pode retornar um erro caso quantidade total de árvores sorteadas seja inferior ao número declarado na função.
A função pode ser customizada de inúmeras maneiras. Utilizei um cálculo genérico para gerar o diâmetro das copas pois não possuo esses registros na base de dados utilizada nesse exemplo, no entanto, altura e diâmetro de copa, se coletados, podem ser utilizados para dimensionar as elipses que representam as copas das árvores. A função geom_ellipse que plota as elipses está implementada no pacote ggforce.
Os nomes científicos são apresentados no gráfico via função geom_label_repel do pacote ggrepel. A diferença entre esta função e a geom_label do ggplot é que a primeira não permite a sobreposição dos labels, facilitando a visualização. Outra opção é plotar um número de identificação ao invés do nome, diminuindo a quantidade de textos no gráfico. Neste caso, seria necessário apresentar, na sequência do gráfico, uma tabela que vincula os números às espécies.